Alternatives Herumgeschnippele
BLOG: Bierologie


 Philipp sagt:
Philipp sagt:
Heute gehts um Alternative Splicing (AS), insbesondere bei Pflanzen.
AS ist mit eine Erklärung für die Proteinvielfalt in allen „höheren“ Organismen. Momentan wird geschätzt das der Mensch über ca. 30.000 Gene, aber 90.000 Proteine verfügt – wie kann das sein?
Die Antwort ist natürlich (wer hätte es bei dem Einstieg auch anders erwartet) Alternative Splicing (Kleine Anmerkung: Trotz Bemühens, für die meisten Fachwörter zwecks Verständnis das deutsche Pendant zu finden – „Alternatives Spleißen“ hört sich einfach zu doof an).
Was passiert also bei AS?
Normalerweise sind Gene bei Eukaryoten so organisiert: Kodierende Regionen werden von unkodierenden flankiert.
In den unkodierenden Regionen verstecken sich u.a. Bindungsstellen für diverse regulierende Genelemente, aber darauf soll hier heute nicht eingegangen werden. Für uns viel wichtiger ist der Aufbau der kodierenden Region; diese unterteilt sich nämlich in Exons und Introns.
Wird im Zellkern die RNA mithilfe der DNA als Kopiervorlage erzeugt (man erinnere sich an die Schule), so bleiben die Introns erstmal drin. Man spricht dann von prä-mRNA. Als nächstes kommt das Spliceosom, ein Batzen aus ca. 300 Proteinen und 5 RNA-Proteinen, welcher sich dieser prä-mRNA annimmt, die Introns heraus spleißt und die Exons miteinander verbindet.
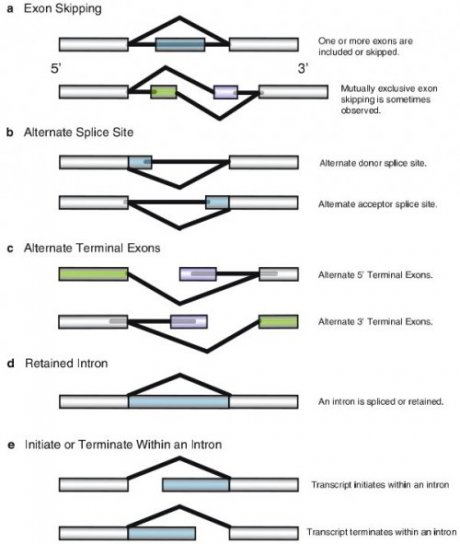
Und hier kommt unser AS ins Spiel, die Introns werden nämlich nicht immer fein säuberlich herausgetrennt, und die Exons bleiben auch nicht immer bis zum Ende dabei. Man unterscheidet momentan 5 Grundtypen von AS;
1. Exon-Skipping (ES): Dabei wird ein Exon übersprungen und kommt nicht mit in die finale mRNA.
2. Alternative Splice Sites: Dabei fängt ein Exon früher an oder hört früher auf.
3. Alternate 5′/3′ Terminal Exons: Die mRNA fängt mit einem anderen Exon an oder hört mit einem anderen auf.
4. Retained Intron: Ein Intron wird in die fertige mRNA mit übernommen.
5. Initiate or Terminate within an Intron: Ein Intron wird nur zum Teil mitgenommen.
Grafik 1 verdeutlicht das ganze nochmal. (Quelle: Haas BJ, Analysis of alternative splicing in plants with bioinformatics tools., Curr Top Microbiol Immunol, Vol 326, p. 17-37)
Diesen Mechanismus kennt man schon seit mindestens 1987 (Breitbart et al. – den Namen hätt ich auch gern, damit kann man spitze Piratenkapitän werden!), und in Menschen wurde er bis dato gut erforscht; so hat man einige Krankheiten auf das Wirken von falschem AS zurückführen können.
Zum Beispiel ist bei Menschen (und den bis dato untersuchten Vertebraten wie Mäusen etc.) Exon-Skipping mit ca. 30% aller AS-Ereignisse am weitesten verbreitet – bei Pflanzen liegt ES bei grade mal 3%.
Man geht davon aus, dass ES zu einer erhöhten Proteindiversität führt – logischerweise kann man so aus einem Bauplan mehrere verschiedene, funktionierende Maschinen bauen.
Interessanterweise ist Intron-Retention (IR) bei Pflanzen am weitesten verbreitet (z.B. bei Wang et al. 2008, Ner-Gaon 2004), und zwar mit ca. 40% aller AS-Ereignisse in den bis jetzt untersuchten Pflanzen (die Prozentzahlen schwanken je nach Veröffentlichung: die Untersuchungen sind abhängig von der verfügbaren Genomsequenz, Anzahl der verfügbaren mRNAs, verwendetem Algorithmus usw.)
Der Unterschied zum Exon-Skipping liegt darin, dass IR das Leseraster verschiebt – kleine Wiederholung:
Die Nukleotide in der RNA sind in Dreiergruppen, sogenannten Tripletts oder Codons, organisiert. Jedes dieser Codons kodiert für eine Aminosäure im fertigen Protein. Wird der Leseraster verschoben, so kommt es zu einer Auflösung des alten Musters, und womöglich kommen in das Protein völlig andere Animnosäuren.
Ein Beispiel: AAU UUA ACU codiert für Asparagin, Leucin und Threonin. Wird der Leserahmen einmal nach rechts verschoben, so erhalten wir A AUU UAA CU, was für Asparagin und ein Stop-Codon codiert. Passiert das in der mRNA, so wird die Translation an dieser Stelle abgebrochen, komme was da noch wolle, und es wird ein verkürztes Protein hergestellt. Meist wird dies von der Zellmaschinerie frühzeitig erkannt, und es kommt zu Non-sense mediated decay – die mRNA wird abgebaut.
Das scheint auch der dahinterliegende Grund in Pflanzen zu sein; durch diese Einrichtung haben Pflanzen eine weitere Stufe in der Kontrolle der Genexpression, die bei uns Tieren dann fehlt, und Pflanzen einen Vorteil in der komplizierten Abstimmung der Proteinmaschinerie gibt. Was allerdings nicht so schlimm ist – wir Tiere können schließlich weglaufen.
Wer sich mit dem Informatikteil dahinter befassen will, dem empfehl ich folgende Veröffentlichungen:
1. Sammeth et al. : A general definition and nomenclature for alternative splicing events. PLoS Comput Biol, Vol 4, Issue 6, 2008.
Es wird eine Nomenklatur vorgestellt, zusammen mit dem Programm „AStalavista“ welches Exongrenzen nimmt, um daran AS zu erkennen. Dann braucht man natürlich noch ein Programm welches die Exongrenzen anhand von ESTs oder mRNAs und einer Genomsequenz ausspuckt! Da gibts z.B. GeneSeqer, GMAP, Splign, Spa usw….
Diese Programme funktionieren alle anders; man muss von Fall zu Fall unterscheiden, welche man nimmt.
Eine Review findet man hier:
2. Barbazuk et al. Genome-wide analyses of alternative splicing in plants: opportunities and challenges., Genome Res, Vol 18, Issue 9, p. 1381-1392, 2008.
Sammeth, M., Foissac, S., & Guigó, R. (2008). A General Definition and Nomenclature for Alternative Splicing Events PLoS Computational Biology, 4 (8) DOI: 10.1371/journal.pcbi.1000147
Barbazuk, W., Fu, Y., & McGinnis, K. (2008). Genome-wide analyses of alternative splicing in plants: Opportunities and challenges Genome Research, 18 (9), 1381-1392 DOI: 10.1101/gr.053678.106