Keine Süßkartoffel ohne Gentechnik

Achtung, ein Thema zum Aufregen! Es geht um grüne Gentechnik. Und ich meine so richtig grüne Gentechnik. Direkt naturgemacht auf den Tisch zu Hause. Ganz ohne Konzern, der damit Profit machen will.
Die meisten gentechnisch veränderten Pflanzen entstehen durch den Gentransfer mit Hilfe bestimmter Bakterien. Dieser Mechanismus ist lange bekannt und wird gerne im Labor für die eigenen Zwecke verwendet. Was wir in diesem Fall Gentechnik nennen, wird in der Natur als ‘Horizontaler Gentransfer’ bezeichnet. Wikipedia weiß dazu, dass es sich dabei um einen Genaustausch zwischen verschiedenen Organsimen handelt, der nicht dem Zweck der Reproduktion dient. Bei Pflanzen wird dieses Phänomen über besagte Bakterien vermittelt, die zur Gattung Agrobacterium zählen. Dabei wird transfer DNA (T-DNA) in Form eines Plasmids in das Genom der Pflanzen eingebaut, was dann je nach Art des Bakteriums unterschiedliche Auswirkungen haben kann.
Der bekannteste Vertreter ist das Agrobacterium tumefaciens. Wie der Name bereits vermuten lässt, verursacht dieses Bakterium in Pflanzen Tumore. Das sieht dann so aus:

Ein weiterer Vertreter, das Agrobacterium rhizogenes, verursacht vermehrte Bildung von Wurzelgewebe. Der eigentliche Sinn des Gentransfers liegt aber woanders. Es werden dabei verschiedene Gene eingebracht, die hauptsächlich der Opin- und Auxinbiosynthese dienen. Die Rolle einer Reihe der übertragenenen Gene ist bisher allerdings noch nicht bekannt. Zur Erzeugung transgener Pflanzen werden diese Sequenzen dann gegen ensprechende Gensequenzen für erhöhten Ertrag, Salztoleranz, Resistenzen etc. ausgetauscht.
Die genetisch veränderten Pflanzen beginnen also Opine zu produzieren. Diese können von der Pflanze selbst nicht verstoffwechselt werden, das Agrobacterium allerdings kann sich davon ernähren. Das schlaue Bakterium hat also eine Pflanze genetisch so verändert, dass sie einen verbesserten Phänotyp besitzt. Dieser wird dann auch an nachfolgende Generationen weitergegeben, auch in Abweseheit von Agrobacterium. Für die Pflanze selbst entsteht daraus allerdings kein Vorteil. Es gibt aber ein prominentes Beispiel, wo diese T-DNA einen positiven Nutzen für Pflanzen hat. Die Rede ist von Nicotiana. Die vorhandenen T-DNA Gene werden abgelesen, allerdings ohne Tumore oder übermäßiges Wurzelwachstum zu verursachen. In der Art Nicotiana glauca wird vermutet, dass das Vorhandensein der T-DNA Auswirkungen auf Entwicklung, sowie Morphologie hat.
In den bisher in der Literatur beschriebenen Fällen sind noch keine essbaren Kulturpflanzen vertreten. Diese Arbeitsgruppe aus Peru (International Potato Center) war gerade dabei die Sequenzdaten der Süßkartoffeln nach sogenannten small interfering RNAs zu durchforsten, als sie dabei auf die Sequenzen der T-DNA gestoßen sind. Eine genauere Analyse zeigte die Anwesenheit eines großen Teils der normalerweise übertragenen Gene an zwei verschiedenen Stellen im Genom. Ob die beiden eingefügten T-DNA Sequenzen von einem oder zwei aufeinanderfolgenden Events verursacht wurden, konnte nicht geklärt werden.
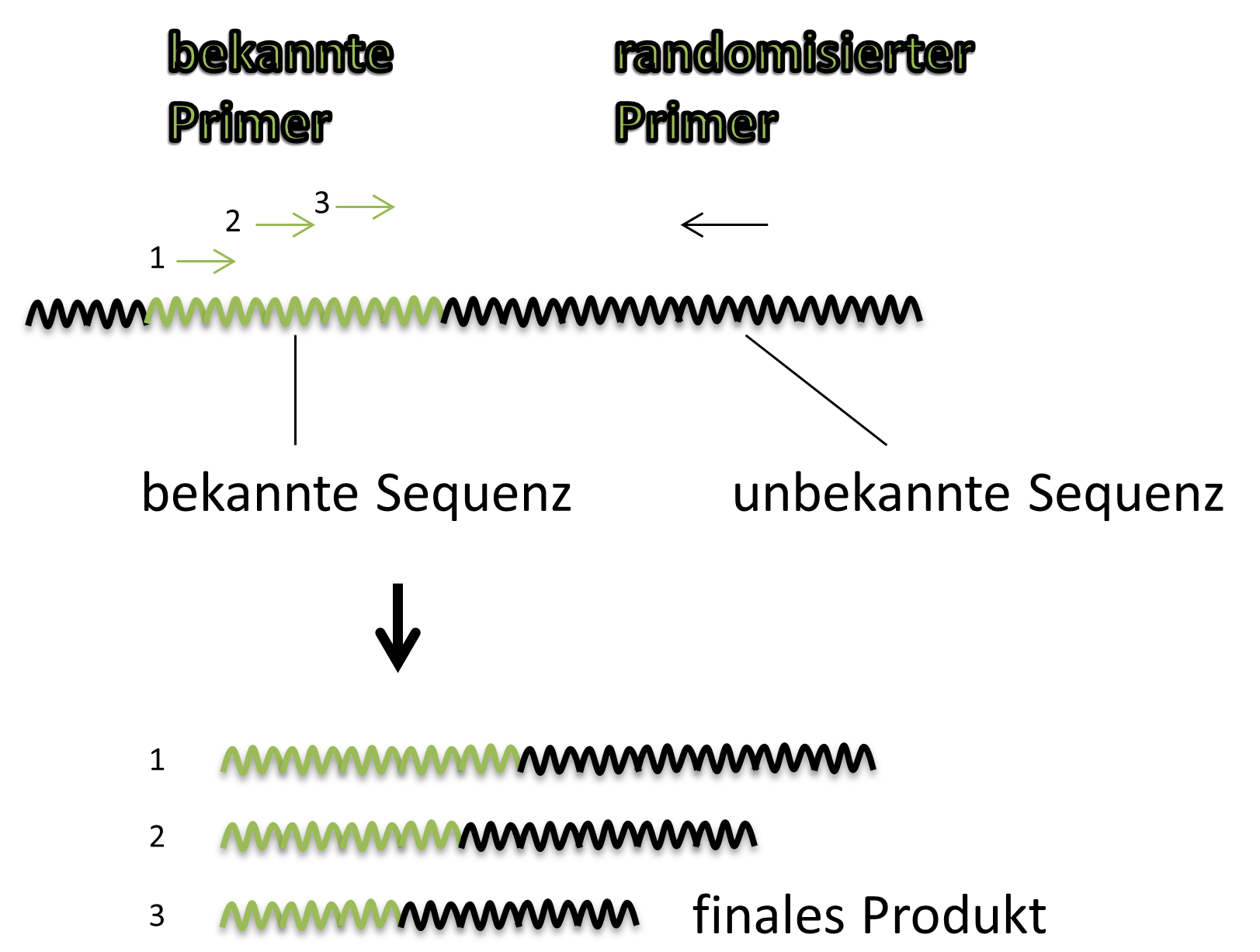
Um herauszufinden, wo genau im Genom sich diese Sequenzen befinden, wurde Genomwalking angewandt. Bei dieser Technik ist die Gensequenz von Interesse bekannt, allerdings ist unklar, wie die sie umgebenden Bereiche aussehen. Um das zu klären, wird eine spezielle Form der DNA-Vervielfältigung verwendet. Um ein Fragment des Genoms zu vervielfältigen benötigt man zwei sogenannte Primer. Das sind kurze Nukleotidsequenzen (ca. 20 Basenpaare), die homolog zu den beiden Enden des zu verfielfältigenden Fragments sind. In diesem Fall muss die Sequenz also vollständig bekannt sein. Beim Genomwalking ist das nicht so. Hier kennen wir nur einen der beiden Primer. Um trotzdem ein Produkt generieren zu können, Bedarf es eines Tricks. Dazu werden in drei aufeinanderfolgenden Vervielfältigungsrunden randomisierte Primer verwendet. Ich versuche es mal an Hand des Bilds unten zu erklären.
In der ersten Runde wird der bekannte Primer 1 und ein randomisierter Primer verwendet. Die exakte Sequenz dieses Primers kennen wir nicht. Er wird an verschiedenen Stellen im Genom binden, u.a. auch außerhalb und in der Nähe unserere bekannten Sequenz. In diesem ersten Schritt werden viele verschiedene Produkte erzeugt werden. Im zweiten Schritt werden diese Produkte als Ausgangsmaterial verwendet. Der bekannte Primer 2 bindet auf einem dieser Produkte und gemeinsam mit randomisierten Primern werden wieder verschiedene Produkte geliefert. Allerdings ist die Anzahl der erzeugten Produkte viel geringer, da weniger Möglichkeiten zur Verfügung stehen. Das Ganze wird noch einmal wiederholt und in der Regel erhält man so ein einziges Produkt oder zumindest einige wenige Produkte. Das gewünschte Produkt kann man dann sequenzieren um so Informationen über umliegende Sequenzen zu gewinnen. Diese Methode nennt man dann übrigens TAIL-PCR.
Durch Vergleich der so gewonnenen Informationen mit verschiedenen Genomen konnte gezeigt werden, dass beide Sequenzen tatsächlich in einem Intron (quasi der stille Teile eines Gens) eines F-Box Gens liegen (kleine Anmerkung: F-Box Gene gehören einer Superfamilie an, die eine bestimmte Struktur aufweisen und am Proteinabbau beteiligt sind). Der Aufbau der Insertionsstelle lässt darauf schließen, dass das Gen vor dem Einbau der T-DNA aktiv war. Eine Expressionsanalyse zeigte dann entsprechend auch, dass die eingefügte T-DNA abgelesen wurde und detektiert werden konnte. Die Analyse zeigte außerdem, dass in Blättern, Wurzel, Stamm und vor allem auch im Speichergewebe T-DNA abgelesen wird. Damit könnten sie also auch eine potenzielle Funktion in der Pflanze besitzen.
Das ist jetzt alles noch nicht besonders interessant. T-DNA Sequenzen lassen sich in einer Vielzahl von Pflanzen nachweisen. Der Vergleich von verschiedenen kultivierten Süßkartoffeln und deren wilden Verwandten zeigte dann aber, dass keine der wilden Süßkartoffeln T-DNA Sequenzen enthielten. Die Autoren argumentieren dann, dass man annehmen kann, dass die T-DNA zur Ausbildung bestimmter Eigenschaften beigetragen hat, die schließlich während der Domestizierung zur weiteren Selektion führten. Da die Süßkartoffel zu einem der häufigsten verzehrten Pflanzen zählt und offensichtlich keine schlechten Einflüsse hat, hoffen die Autoren weiterhin, dass die öffentliche Meinung über ‘unnatürliche’ transgene Pflanzen so positiv beeinflusst werden kann. Da diese Debatte ja eher von Emotionen als von Fakten geprägt ist, kann ich mir das leider nur schwer vorstellen. Aber ich wünsche den Autoren viel Erfolg beim Verbreiten dieser Botschaft.
Merken


Kurze technische Frage: ist “der” randomisierte Primer irgendein Primer mit einer festen, aber beliebigen Sequenz oder ein Primer-Mix, der Primer mit verschiedenen Sequenzen enthält?
Beides stimmt ein bisschen. Die Sequenz eines randomisierten Primers ist einigermaßen fest vorgegeben, insofern als das bestimmte Positionen “fest” sind. Bei anderen Positionen ist es egal welches Nukleotid eingebaut wird, bzw. entweder A/T, C/G etc. Nach dem DNA IUPAC 1-letter code, könnte ein randomisierter Primer dann wie folgt aussehen: NGTCGASWGANAWGAA. Kann man direkt so bestellen. In der Summe bekommt man dann ein wildes Durcheinander an verschiedenen Primern.
Ah, okay. Danke für die Aufklärung (und den Artikel!).
Die Süsskartoffel als von Haus aus transgene Pflanze hat das GM-Verbot also unterlaufen bevor es überhaupt existierte.
Bedeutet aber die Existenz von natürlichen oder schon lange kultivierten transgenen Pflanzen, dass Genmodifikation von Pflanzen generell unproblematisch ist? Nicht unbedingt, denn der (horizontale) Gentransfer in der Natur ist wohl um Grössenordnungen langsamer als der Gentransfer durch menschliche Eingriffe. Was die Natur in Jahrhunderten schafft, könnte der Mensch in Jahrzehnten schaffen. Nur eins lässt sich mit Bestimmheit sagen: Prinzipiell ist der Gentransfer über Artengrenzen hinweg nichts “unnatürliches” und nichts von vornherein schädliches. Das heisst allerdings nicht, dass es in jedem Fall unproblematisch ist. Da jede Genmodifikation bei Pflanzen ausgedehnte Testbatterien durchlaufen muss, gilt dennoch, dass die Wirkung von GM-Food auf die menschliche Gesundheit und die Auswirkungen auf die Umwelt wohl besser untersucht und kontrolliert ist als es die Auswirkungen der meisten konventionellen Züchtungen sind.
“Ganz ohne Konzern, der damit Profit machen will.”
Wie die Leute aber auch auf solche lächerlichen Ideen kommen! Wenn Sie sich schon so plump an die “gemeinnützigen” Konzerne ranwanzen, dann schreiben Sie doch wenigstens Disclaimer davor.
Das gleiche gilt für den emotionalen Schluß.
Pingback:Lila Tomaten » Von Menschen und Mäusen » SciLogs - Wissenschaftsblogs